| Les chercheurs du Laboratoire de Physique des Solides (Université Paris-Saclay/CNRS) et du Laboratoire Lumière, Matière et Interface (Université Paris-Saclay/ENS Paris-Saclay/CNRS/Centrale Supélec) ont mis au point une méthode expérimentale pour visualiser en temps réel la croissance de virus sphériques individuels en utilisant la microscopie de fluorescence en réflexion totale interne. |
Tous les virus sur Terre exploitent la machinerie cellulaire de leur hôte pour se répliquer. Une des étapes cruciales du cycle de vie d’un virus met en œuvre un processus complexe d’auto-assemblage, qui a lieu malgré l’encombrement macromoléculaire et l’activité enzymatique intense régnant dans le cytoplasme. Les virus sphériques les plus simples s’auto-assemblent par croissance stochastique de leur coque de protéines—appelée capside—autour de leur génome constitué d’une, ou parfois plusieurs, chaines d’ARN.
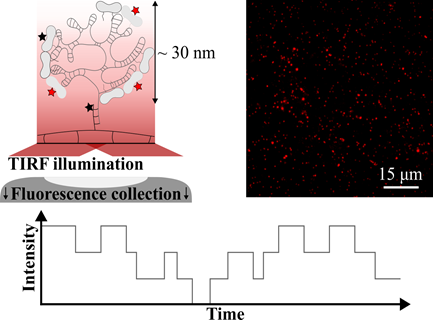
Dans cette étude, les chercheurs du Laboratoire de Physique des Solides et leurs collaborateurs ont implémenté une méthode expérimentale pour sonder la dynamique de croissance des virus à l’échelle des protéines individuelles. La méthode repose sur l’utilisation de la microscopie de fluorescence à réflexion totale interne (TIRF) pour suivre l’évolution de l’intensité lumineuse de centaines de sites d’assemblage simultanément à mesure que des protéines fluorescentes s’y accrochent ou au contraire s’en détachent. Grâce à un algorithme de détection de marches combiné à une analyse statistique, les chercheurs ont réussi à estimer des grandeurs microscopiques telles que le taux d’accrochage et le temps de résidence moyen, qui sont inaccessibles par des techniques traditionnelles de moyenne d’ensemble.
L’imagerie de fluorescence en molécule unique se révélera cruciale pour élucider l’auto-assemblage de virus au niveau moléculaire, particulièrement dans des milieux encombrés et actifs mimant les conditions cytoplasmiques. Ces résultats sont publiés dans la revue scientifique Nano Letters.
Contributeur.ice.s :
Équipe Structure et Dynamique d’Objets Biologiques Auto-Oganisés (SOBIO)
Laboratoire Lumière, Matière et Interface (LuMIn)
Centre Borelli
Financements :
LabEx PALM et NanoSaclay
Références : T. Bugea, R. Suss, L. Gargowitsch, C. Truong, K. Perronet, G. Tresset, Nano Lett. 24 (2024) 14821-14828. https://doi.org/10.1021/acs.nanolett.4c04458
Contact :
guillaume.tresset@universite-paris-saclay.fr
